影像组学在肺癌精准医学中的应用
前言
肺癌是世界上最具侵袭性的人类癌症之一,其5年总体生存率为10%~15%,在过去的30年里没有明显的改善[1-2]。总共有87%的肺癌被诊断为非小细胞肺癌(NSCLC),其组织学类型包括腺癌、鳞状细胞癌和大细胞癌。肺癌根据分子亚型进行分类,基于特定的基因改变而驱动和维系肺肿瘤发生[3]。这样的驱动基因突变,以及与基因突变相关的突变信号蛋白的持续活跃,对肿瘤细胞的生存至关重要,并促进了新的靶向治疗的发展[4]。目前,对于不可切除的NSCLC的标准治疗方案是同步放化疗,但有一半以上的患者局部治疗失败[5]。最新证据表明,治疗前和治疗后解剖或功能/分子成像信息可以用来制订治疗的类型和强度,并预测放疗的治疗结果。例如,计算机断层扫描(computed tomography,CT)上的肿瘤体积改变已经被用于预测NSCLC患者的放疗反应[6-7]。功能/分子成像,特别是正电子发射断层显像(positron emission tomography,PET)的18F-氟代脱氧葡萄糖(fludeoxyglucose,FDG)成像,作为预测放疗效果的潜在预后因素已受到特别关注[8],并已被证明有助于圈定Ⅲ期NSCLC[9-10]的照射野。例如,对77例适度调强放疗(平均放疗剂量为66 Gy)的Ⅲ期NSCLC患者,检测放疗前和放疗中期的FDG-PET/CT指标的疗效预测价值[11]。一些放疗前和放疗中期的PET指标被发现是局部复发和区域/远处复发的先兆,但不能预测总生存时间。
传递和成像技术的进步使图像引导和自适应放射治疗(image-guided and adaptive radiotherapy,IGART)跨向了新时代。IGART技术见证了人们对于应用不同成像方式的兴趣,包括定义靶区容积或者预测治疗反应。现代的IGART在肿瘤分期和优化不同癌症类型的治疗规划时,医生对利用多模型成像有着浓厚的兴趣[12]。目标是通过结合互补的解剖信息(CT、MRI、超声等)改进的靶区定义,外加通过功能性和分子成像(PET,多功能MRI等)改善疾病特征和定位。最近,以治疗中的FDG-PET定义肿瘤对剂量升高适应性治疗的靶区容积变化,显示应用高剂量放疗(最高总剂量为86 Gy)治疗局部Ⅰ~Ⅲ期NSCLC肿瘤有良好的治疗反应,放疗诱导毒性发生率也可以接受[13]。本组试验中的42例患者达到了主要目标即提高2年局部肿瘤控制率,其中中央型肿瘤控制率为82%,整体肿瘤控制率为62%,5年总体生存率为30%[13]。此外,Ⅲ期NSCLC患者在放疗中应用FDG-PET/CT,发现在没有高剂量放疗的情况下,2年局部复发累积发生率约为24%[11],而RTOG 0617试验的发生率约为34%[14]。
在放射肿瘤学中,来自影像数据的信息与治疗终点有关,尽管将提取相关特征为某一特定任务仍然具有挑战性,且尚未完全探明。正如之前提出的[15],这个问题可以根据一种工程学的“模式识别”方法来解决,而这需要了解临床终点和成像方式的特征。这个从解剖/分子图像中提取出相应的生物信息和临床终点的定量信息的过程是一个被称为“影像组学”的新兴领域[16-17]。可以认为影像组学包括两个主要步骤:①从先前定义的肿瘤区域提取定量成像的静态和动态特征;②然后将成像特征纳入数学模型中,用于治疗结果的预测,旨在与常用的临床预测因素进行比较,为治疗方案的个体化提供附加价值。
NSCLC一直处于放射组学研究的最前沿,比如以CT为基础的三个放射学特征可以预测肺腺癌的远处转移[18]。采用Relief特征选择法和Naïve Bayes分类器,从治疗前的CT图像中提取信息预测组织学的亚型(腺癌或鳞状细胞癌),AUC为0.72[19]。结合图像影像组学和临床特征,提高了对无病生存的预测[20]。与PET/CT的结合可以预测局部控制[21]。影像组学特征的变化(δ影像组学)被证明可以预示NSCLC的预后[22]。Avanzo等对放疗的影像组学进行了综述[23]。在接下来的研究中,我们描述了使用单一和/或多模态成像数据进行肺癌影像组学分析的力学。
方法
图像分割
医学图像分割是一种从背景或其他相邻结构中的图像分离出感兴趣的结构的过程。这是许多医学成像应用(包括影像组学)的必要前提步骤。有一些商业和学术软件工具支持不同的分割算法。一般来说,商业软件包有更好的操作,比如用户容易熟悉的界面以手动和半自动的分割,但往往落后于该领域的最新发展。相比之下,学术软件包,如ITK[24]、BioImage Suite[25]、MIPAV[26]和ImageJ[27],3D切片机[28]可能倾向于面向单模式应用,在需要处理多模态图像时就显得不太容易操作。这些算法属于不同的类别,例如,Pham及其同事将细分算法分为8个不同的类别:阈值、区域增长、分类器、聚类、Markov随机字段模型、人工神经网络、可变形模型和atlas-引导方法[29]。最粗暴的图像分割是基于变形模型(蛇或水平集),它是影像域中显性或隐性定义的曲线或平面的几何代表。这些模型在内部因素(轮廓曲率)和外部因素(图像边界限制)的影响下活动[30-31]。图1显示了一个使用多值水平集算法显示肺癌中PET/CT联合分割的例子[32]。
图像特征提取
从图像中提取的特征,根据扫描时的参数获取可分为静态(在一个时间点的增强快照)和动态(时间变量)特征,根据扫描节点分为治疗前及治疗过程中的特征[33]。静态特性都是基于强度、物体形态和纹理,如我们之前关于PET图像模式识别分析的工作[15]或我们在乳房X光检查数据库基于内容检索的相似性学习[34-35]。动态特征是从时变采集中提取的,如动态PET,SPECT或MR。这些特征基于使用组织室间模型的动力学分析以及与运输和结合率有关的参数[36]。
静态图像的特性
几个静态图像特征可以应用于影像组学研究。
PET的描述符:标准摄取值(standard uptake value,SUV)
放射示踪强度值是提取的PET图像,并将其转化为具有统计描述符的SUVs,如最大值、最小值、均值、标准差(standard deviation,SD)和变异系数(coefficient of variation,CV)。SUV是PET图像定量分析的一种标准方法[37],类似于从CT Hounsfield单位(HU)中导出类似的指标或者在从MRI中总结强度统计。
强度体积直方图(intensity volume histogram,IVH)
IVH的定义方式与剂量体积直方图(dose volume histogram,DVH)相似:IVH上的每个点将该结构的绝对或相对体积定义为最大强度的百分比超过可变强度阈值。因此,IVH旨在发挥与DVH相同的作用,将复杂的3D数据缩减为一个更简单的直方图来解释。这种方法将允许从功能图像中提取若干指标用于结果分析,比如Ix(最小强度到x%的最高强度体积),Vx(体积百分比含最少的x%强度值),和描述性统计(均值、最小值、最大值、标准差等)。
纹理特征
在不同的研究中使用几种的纹理特征,比如使用灰度共生矩阵(gray-level co-occurrence matrices,GLCM),邻域灰度差分矩阵(neighborhood gray-tone difference matrices,NGTDM),灰度运行长度矩阵(gray-level run length matrices,GLRLM)和灰度级大小区域矩阵(gray-level size zone matrices,GLSZM)的纹理特征,或者Laws能量测量[38-39]。在这里,我们简要描述GLCM特征,它是最常用的纹理之一。这个矩阵表示定义强度级别(由i和j索引)的次数出现在整个3D图像中的相邻立体像素之间。从数学上讲,这可以看作是给定纹理图像的二阶联合条件概率密度函数的一个扩展。对于有M强度箱的图像,共现矩阵大小为M×M。M等级是通过应用图像量化方法限制矩阵的大小而获得。M的典型值是以2的幂次来选择(8、16、32、64等)。应该强调的是,这些指标与肿瘤的位置、方向、大小和亮度无关,并考虑到局部强度-空间分布[40-41]。与直接(一阶)直方图度量(例如均值和标准差)相比,其仅测量与肿瘤微环境中的与空间分布无关的强度可变性,这是一个重要的优势。
动态图像特征
为了量化一个对比剂或在邻域(可以是一个或多个体素)基础上的示踪剂动态行为,隔室模型方法通常用于动态成像[36]。作为一个例子,我们简要回顾了用于FDG-PET的隔室模型的特征,并将类似的原理应用于动态CT或MRI。
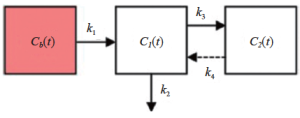
FDG的动力学模型[36]。用3室模型描述了FDG-6-磷酸(FDG6P)在肿瘤中的诱捕作用。在图2中,Cb(t)表示输入函数;C1(t)为非磷酸化FDG的浓度;C2(t)为FDG-6-磷酸的浓度。通过GLUTs跨膜的双向转运由速率常数k1和k2表示,FDG的磷酸化由k3表示,而G6-磷酸酶的作用以速率常数k4表示。我们将开始分析,假设k4=0,但我们将探索非零k4。通过对隔室模型的评估,通过KFDG=k1*k3 /(k2+k3)的关系对FDG摄取速率(K)的测量值进行评估,我们期望邻域K值的统计属性能够预测局部控制。
多度量建模的响应
在数据驱动结果建模的背景下,观察到的治疗结果被认为是由多重剂量、临床和生物输入变量引起的[42]。放射肿瘤学的结果包括两个指标:肿瘤控制概率(tumor control probability,TCP)和周围正常组织并发症概率(normal tissue complication probability,NTCP)[43-44]。在结果建模之前,特征选择将被用于为模型找到最佳的特征子集。特征选择工作主要有三种技术:过滤方法、包装方法和嵌入式方法[45]。基于滤波的方法是一种计算效率高的基于信息的方法,包括交互信息、基于关联和救济方法。包装方法使用分类器的预测性能来评估特征的重要性,而嵌入式方法在训练过程中实现特征的选择。通过优化选择的特性,可以建立一个数据驱动模型,如经典的逻辑回归方法或更先进的机器学习技术。
关于放射治疗结果建模的更多细节,读者可以参考我们以前的工作[46]和剂量反应探测系统(dose-response explorer system,DREES),后者是目的导向的专用软件工具[47]。下面,我们将展示使用标准逻辑回归和更先进的机器学习的例子。
逻辑回归的结果建模
逻辑建模是多度量建模的常用工具。在我们以前的工作中[42,48],使用一个分对数变换:
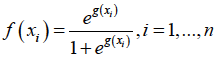
其中n为病例数(患者),xi为输入变量值的向量(例如图像特征)用来预测yi的结果f(xi)(例如ith的TCP或NTCP)。
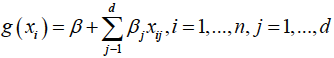
其中d为模型变量的数量,而β’s是通过最大化数据产生观察结果的概率确定的一组模型系数。参数的数量可以从特征选择方法或合并所有特征和应用收缩方法(如LASSO)来确定[49]。
通过机器学习进行结果建模
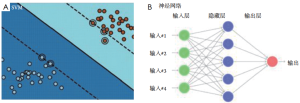
机器学习代表广泛的人工智能技术,比如神经网络、决策树、支持向量机(support vector machines,SVM)。这些技术能够通过从给定的输入数据中学习周围环境来模拟生物的智能。基本上,SVM[50]和神经网络都是监督学习(一般在基于图像的结果建模中应用),旨在估计来自已知(输入、输出)样本的未知(输入、输出)映射。这些方法越来越多地用于放射肿瘤学,因为它们能够检测数据中的非线性模式[51]。特别是,神经网络被广泛研究以模拟肺损伤病例的放疗后治疗结果[52-53]。在这一背景下,学习被定义为从数据中估算依赖关系[54]。
从输入数据和标签yi,N个总样本的D={(xi,yi) ∈ Rn×C | i=1,2,3,...N},可以找到函数f: Rn→C,对于每个样本i的f(xi) ≅ yi。两种算法可以遇到:分类,目标集合C被离散化;或者回归,当C=R时。例如,以C={1,−1}为代表的二元分类(例如肿瘤治疗失败的低/高风险),SVM将在一些更高维度上寻找一个正在发展的超平面,这样数据点就会根据其标签(图3A)与最大的边界分开。在数学上,SVM由优化问题描述:
Maxw,b,rr和,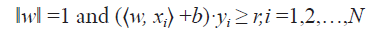
其中,r ∈ R,w ∈ Rn和yi={1,−1}是要优化的变量。从本质上讲,目标函数r是被发现的最大边界,强加约束以使得每个数据点(xi,yi)的下界约束在决策边界上,至少大于r。因此,最优权重(w,b)是SVM二进制分类器的唯一决定变量:
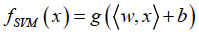
其中,指标函数的定义为: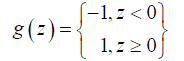
另一方面,神经网络在矩阵(权值)的集合中使用自迭代构造了一个非线性函数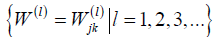 和向量(偏倚)
和向量(偏倚)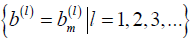 。
。
比如:
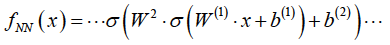
式4是一种被称为激活的非线性函数,它在神经网络中起着重要的作用。从(d)中,权值和偏倚的集合决定了神经网络函数fNN,而非线性则被理解为仅仅来自于激活函数。同样,这里的“W[1],W[2],…”表示权重的矩阵形式,与偏置向量“b[1],b[2],…”相同。指数l=1,2,3,...表示权重和偏差的深度称为层(图3B)。一个浅层网络(层数小)可以表示一个非常复杂的函数,只要神经元的数目足够大,并且在层中越深,神经网络所代表的功能就越复杂。在监测下,例如,实施fnn(xi) ≅ yi,然后给最好的权重和偏见 将被利用。
将被利用。
在所有的机器学习技术中,深度神经网络最近在各种生物医学问题上表现出令人印象深刻的表现,如患者分类、治疗计划和理解生物过程。这些方法不需要详细地提取上述讨论的特征。例如,Kuruvilla等利用从分段肺中提取的静态特征与前馈和前馈反向传播网络进行肺癌分类[55]。如Paulin等[56]所提出的,他们使用13个训练函数(反向传播神经网络)进行分类。该方法进行肿瘤分类的准确度为93.3%,特异性为100%,敏感性为91.4%[55]。此外,Kumar等提出使用自编码器在CT图像中提取的深层特征,其基于神经网络将数据“编码”到一个潜在空间,连同一个作为肺癌分类器的二元决策树(例如,恶性 vs 良性)分类[57]。在输出层面上,利用“解码”变换进行数据重建,获取被提取并应用到训练分类器的学习特征。
肺癌的放射学应用实例
利用CT成像对患者生存建模
Aerts等[58]建立由4个特征组成的放射学特征,“统计能量”描述肿瘤的整体密度,“形状紧凑度”量化肿瘤形状有多么紧凑,“灰度级不均匀性”用于度量异质性,以及小波“灰度非均匀性HLH”也描述了中频分解图像后的肿瘤内异质性。通过使用RIDER数据集选择最稳定的特征来获得特征,然后基于422例肺癌患者的信息进行训练,以预测生存。在一个独立的肺部数据集上进行评估的特征可以预测c指数(c-index,CI)为0.65的存活率,并成功地测试了不同类型的癌症(肺癌、头颈癌和颈癌)的队列,从而证明了影像组学在不同癌症之间的转化能力。在同一项研究中,对来自肺癌队列的89名患者的基因表达量进行了21 766个基因的测定,并揭示了在放射学特征和基因表达模式之间的重要联系。
利用PET成像对患者生存建模
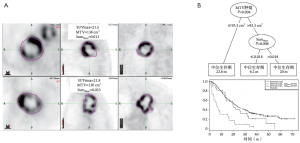
Ohri等[59]最近发表了一个拥有201例患者的多中心数据的辐射组学模型。通过使用LASSO程序,他们确定了从GLCM(SumMean)计算出的一个纹理特征,SumMean可作为总体生存的独立预测因子,补充了决策树中的代谢肿瘤体积(metabolic tumor volume,MTV)。在决策树中,对93.3 cm3以上的肿瘤,最优的SumMean阈值为0.018(图4)。
利用PET/CT对肿瘤反应建模
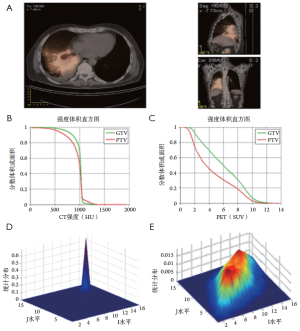
将PET/CT或PET/MRI等多种样式的影像学信息结合在一起是一种固有的优势。在对30例NSCLC患者的回顾性研究中,从PET和CT图像中提取了30个特征,包括有动作校正及无动作校正,如图5所示[33]。这些特征包括肿瘤体积、SUV/HU值(如平均值、最小值、最大值和标准差)、IVH指标以及基于纹理的特征(如能量、对比度、局部均匀性和熵)。使用基于四种4D-CT数据集的反卷积方法,基于总体平均概率扩散函数(probability spread function,PSF)修正运动伪影的数据[60]。图5显示了这种特征的一个例子。然后,这些特征可以通过DREES软件实现(图6),以预测局部故障[21],该模型由基于IVHs的PET和CT两个参数的模型组成,提供了最佳性能。
放射基因组学实例
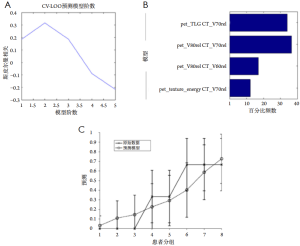
成像信息除了与临床终点相关,还可能与生物学和基因分子特征有关。在NSCLC中发现表皮生长因子受体(epidermal growth factor receptor,EGFR)突变的患者是非常意义的,因为他们可能对EGFR靶向药物有反应,且用于检测EGFR突变的分子方法非常昂贵。治疗前CT扫描的放射学特征能量定律(laws-energy),对EGFR突变状态有重要的预测作用。通过多重Logistic回归和配对选择,建立了一种基于种子区域生长方法的肿瘤分割半自动识别EGFR突变状态的放射学模型,有中等预测价值(AUC,0.647;95%Cl:0.576~0.701)。该模型若引入其他变量,如临床变量,可将AUC提高到0.709[61]。图7显示了一个实例。基因融合已成为临床研究的热点,因为靶向药物可以使患者受益。
讨论
近年来,在放射治疗反应的结果建模中使用成像技术的应用迅速增加,见证了现有的癌症治疗成像技术在常规治疗特别是放射治疗中应用价值的提高。然而,目前有几个问题限制了它的快速发展。图像采集参数可能会影响提取特征的重现性,从而影响这些特征对治疗预测的稳健性和稳定性。这包括静态特性,例如SUV/HU/MRI描述符和纹理特征。有趣的是,基于纹理的特征被证明具有与简单的SUV描述符相似或更好的重现性[62]。这就要求标准化的获取参数。此外,影响这些特征稳定性的因素还包括信噪比(signal-to-noise ratio,SNR)、部分体积效应、运动伪影、参数设置、重复采样大小及图像量化[15,63]。事实上,一个3D(静态)PET可能无法提供一个移动肿瘤的精确位置、体积和吸收的活动分布。将传统的(3D)PET/CT与呼吸门控(4D)PET/CT、PET进行比较,以评估呼吸运动对模型[64]和肺癌患者[65]中提取的特征变异性的影响。在吸气/呼气时,三维图像与4D图像之间的长轴长度不一致,表明肿瘤的形状和旋转在不同的时像之间存在差异[65]。然而,硬件和软件技术的进步将促进高级图像处理技术在医学成像领域的广泛应用,以达到更好的临床效果。例如,预处理方法(如去噪和反卷积方法)已经有助于减少这种伪影[66-67],然而,基于非局域性和稀疏性的更高级的图像恢复方法可能更有效[68]。使用逻辑回归的结果建模已经成为一个事实上的标准,然而,更高级的建模技术,如机器学习,可以提供进一步的预测能力,特别是在处理更复杂和非线性关系的特征和临床结果之间。我们相信,图像分析与机器学习[51]之间的协同作用可以提供有力的工具来加强和进一步利用临床实践中基于图像的结果建模,以改善未来的临床决策和个体化医疗。
结论
在这篇综述中,我们概述了影像组学在肺癌中的应用,从基本的图像分割、结果建模,到破译遗传特征。随着成像技术在肺癌管理中的作用不断增强,影像组学也将随之而起。然而,人们认识到,这一领域仍处于起步阶段,目前面临着诸如呼吸运动伪影的图像参数标准化等挑战,可能限制了其可重复性和在临床试验中的应用。但是,研究和临床领域的共同努力旨在克服这些挑战,并从个体化精准医疗影像组学的潜力中获益。
Acknowledgements
None.
Footnote
Conflicts of Interest: The authors have no conflicts of interest to declare.
References
- Stewart BW, Wild CP. World Cancer Report 2014 [Internet]. Cited 2017 Sep 1. Available online: http://publications.iarc.fr/Non-Series-Publications/World-Cancer-Reports/World-Cancer-Report-2014
- Jemal A, Siegel R, Ward E, et al. Cancer statistics, 2008. CA Cancer J Clin 2008;58:71-96. [Crossref] [PubMed]
- Pao W, Girard N. New driver mutations in non-small-cell lung cancer. Lancet Oncol 2011;12:175-80. [Crossref] [PubMed]
- Travis WD, Brambilla E, Riely GJ. New pathologic classification of lung cancer: relevance for clinical practice and clinical trials. J Clin Oncol 2013;31:992-1001. [Crossref] [PubMed]
- Blackstock AW, Govindan R. Definitive Chemoradiation for the Treatment of Locally Advanced Non-Small-Cell Lung Cancer. J Clin Oncol 2007;25:4146-52. [Crossref] [PubMed]
- Seibert RM, Ramsey CR, Hines JW, et al. A model for predicting lung cancer response to therapy. Int J Radiat Oncol Biol Phys 2007;67:601-9. [Crossref] [PubMed]
- Ramsey CR, Langen KM, Kupelian PA, et al. A technique for adaptive image-guided helical tomotherapy for lung cancer. Int J Radiat Oncol Biol Phys 2006;64:1237-44. [Crossref] [PubMed]
- de Geus-Oei LF, Oyen WJ. Predictive and prognostic value of FDG-PET. Cancer Imaging 2008;8:70-80. [Crossref] [PubMed]
- Konert T, Vogel W, MacManus MP, et al. PET/CT imaging for target volume delineation in curative intent radiotherapy of non-small cell lung cancer: IAEA consensus report 2014. Radiother Oncol 2015;116:27-34. [Crossref] [PubMed]
- Hallqvist A, Alverbratt C, Strandell A, et al. Positron emission tomography and computed tomographic imaging (PET/CT) for dose planning purposes of thoracic radiation with curative intent in lung cancer patients: A systematic review and meta-analysis. Radiother Oncol 2017;123:71-7. [Crossref] [PubMed]
- Gensheimer MF, Hong JC, Chang-Halpenny C, et al. Mid-radiotherapy PET/CT for prognostication and detection of early progression in patients with stage III non-small cell lung cancer. Radiother Oncol 2017. [Epub ahead of print]. [Crossref] [PubMed]
- Jaffray DA. Image-guided radiotherapy: from current concept to future perspectives. Nat Rev Clin Oncol 2012;9:688-99. [Crossref] [PubMed]
- Kong F-M, Ten Haken RK, Schipper M, et al. Effect of Midtreatment PET/CT-Adapted Radiation Therapy With Concurrent Chemotherapy in Patients With Locally Advanced Non-Small-Cell Lung Cancer: A Phase 2 Clinical Trial. JAMA Oncol 2017. [Epub ahead of print]. [Crossref] [PubMed]
- Bradley JD, Paulus R, Komaki R, et al. Standard-dose versus high-dose conformal radiotherapy with concurrent and consolidation carboplatin plus paclitaxel with or without cetuximab for patients with stage IIIA or IIIB non-small-cell lung cancer (RTOG 0617): a randomised, two-by-two factorial phase 3 study. Lancet Oncol 2015;16:187-99. [Crossref] [PubMed]
- El Naqa I, Grigsby P, Apte A, et al. Exploring feature-based approaches in PET images for predicting cancer treatment outcomes. Pattern Recognit 2009;42:1162-71. [Crossref] [PubMed]
- Lambin P, Rios-Velazquez E, Leijenaar R, et al. Radiomics: Extracting more information from medical images using advanced feature analysis. Eur J Cancer 2012;48:441-6. [Crossref] [PubMed]
- Kumar V, Gu Y, Basu S, et al. Radiomics: the process and the challenges. Magnetic Resonance Imaging 2012;30:1234-48. [Crossref] [PubMed]
- Coroller TP, Grossmann P, Hou Y, et al. CT-based radiomic signature predicts distant metastasis in lung adenocarcinoma. Radiother Oncol 2015;114:345-50. [Crossref] [PubMed]
- Wu W, Parmar C, Grossmann P, et al. Exploratory Study to Identify Radiomics Classifiers for Lung Cancer Histology. Front Oncol 2016;6:71. [Crossref] [PubMed]
- Huang Y, Liu Z, He L, et al. Radiomics Signature: A Potential Biomarker for the Prediction of Disease-Free Survival in Early-Stage (I or II) Non-Small Cell Lung Cancer. Radiology 2016;281:947-57. [Crossref] [PubMed]
- Vaidya M, Creach KM, Frye J, et al. Combined PET/CT image characteristics for radiotherapy tumor response in lung cancer. Radiother Oncol 2012;102:239-45. [Crossref] [PubMed]
- Fave X, Zhang L, Yang J, et al. Using Pretreatment Radiomics and Delta-Radiomics Features to Predict Non-Small Cell Lung Cancer Patient Outcomes. Int J Radiat Oncol Biol Phys 2017;98:249. [Crossref] [PubMed]
- Avanzo M, Stancanello J, El Naqa I. Beyond imaging: The promise of radiomics. Phys Med 2017;38:122-39. [Crossref] [PubMed]
- Yoo TS, Ackerman MJ, Lorensen WE, et al. Engineering and algorithm design for an image processing Api: a technical report on ITK--the Insight Toolkit. Stud Health Technol Inform 2002;85:586-92. [PubMed]
- Papademetris X, Jackowski MP, Rajeevan N, et al. BioImage Suite: An integrated medical image analysis suite: An update. Insight J 2006;2006:209. [PubMed]
- McAuliffe MJ, Lalonde FM, McGarry D, et al. Medical Image Processing, Analysis and Visualization in clinical research. 14th IEEE Symposium on Computer-Based Medical Systems. Bethesda, MD, 2001:381-6. Available online: http://ieeexplore.ieee.org/document/941749/
- Abramoff MD, Magalhães PJ, Ram SJ. Image processing with ImageJ. Biophotonics International 2004;11:36-42.
- Fedorov A, Beichel R, Kalpathy-Cramer J, et al. 3D Slicer as an Image Computing Platform for the Quantitative Imaging Network. Magn Reson Imaging 2012;30:1323-41. [Crossref] [PubMed]
- Pham DL, Xu C, Prince JL. Current Methods in Medical Image Segmentation. Annu Rev Biomed Eng 2000;2:315-37. [Crossref] [PubMed]
- Sethian JA. Level Set Methods and Fast Marching Methods: Evolving Interfaces in Computational Geometry, Fluid Mechanics, Computer Vision, and Materials Science. Cambridge: Cambridge University Press, 1999:402.
- Xu C, Pham D, Prince J. Image Segmentation Using Deformable Models. In: Sonka M, Fitzpatrick J. editors. Handbook of Medical Imaging. Medical Image Processing and Analysis. SPIE Press, 2000:129-74.
- El Naqa I, Yang D, Apte A, et al. Concurrent multimodality image segmentation by active contours for radiotherapy treatment planning. Med Phys 2007;34:4738-49. [Crossref] [PubMed]
- Naqa IE. The role of quantitative PET in predicting cancer treatment outcomes. Clin Transl Imaging 2014;2:305-20. [Crossref]
- El Naqa I, Wei L, Yang Y. Content-based image retrieval for digital mammography. In: Mohammed S, Fiaidhi J. editors. The Ubiquity 2.0 Trend and Beyond. Hershey: IGI Global, 2010:485-508.
- El-Naqa I, Yang Y, Galatsanos NP, et al. A similarity learning approach to content-based image retrieval: application to digital mammography. IEEE Trans Med Imaging 2004;23:1233-44. [Crossref] [PubMed]
- Watabe H, Ikoma Y, Kimura Y, et al. PET kinetic analysis--compartmental model. Ann Nucl Med 2006;20:583-8. [Crossref] [PubMed]
- Strauss LG, Conti PS. The applications of PET in clinical oncology. J Nucl Med 1991;32:623-648; discussion 649-650. [PubMed]
- Khalil MM. editor. Basic Science of PET Imaging. Springer, 2017. Available online: http://link.springer.com/content/pdf/10.1007/978-3-319-40070-9.pdf
- Vallières M, Freeman CR, Skamene SR, et al. A radiomics model from joint FDG-PET and MRI texture features for the prediction of lung metastases in soft-tissue sarcomas of the extremities. Phys Med Biol 2015;60:5471. [Crossref] [PubMed]
- Castleman KR. editor. Digital Image Processing. New Jersey: Prentice Hall Inc., 1979.
- Haralick RM, Shanmugam K, Dinstein I. Textural Features for Image Classification. IEEE Transactions on Systems, Man, and Cybernetics 1973;SMC-3:610-21. [Crossref]
- Deasy J, El Naqa I. Image-Based Modeling of Normal Tissue Complication Probability for Radiation Therapy. In: Bentzen SM, Harari PM, Mackie TR, et al. editors. Radiation Oncology Advances. Springer, 2007.
- Steel GG. editor. Basic clinical radiobiology. 3rd ed. London: Arnold; New York: Oxford University Press, 2002. Available online: http://trove.nla.gov.au/version/45382700
- Webb SP. editor. The physics of three-dimensional radiation therapy conformal radiotherapy, radiosurgery, and treatment planning. Bristol: UK Institute of Physics Pub, 2001. Available online: http://trove.nla.gov.au/work/10354339
- Guyon I, Elisseeff A. An introduction to variable and feature selection. J Mach Learn Res 2003;3:1157-82.
- El Naqa I. Outcomes Modeling. In: Starkschall G, Alfredo R, Siochi C. editors. Informatics in Radiation Oncology. CRC Press, 2013:257-75.
- El Naqa I, Suneja G, Lindsay PE, et al. Dose response explorer: an integrated open-source tool for exploring and modelling radiotherapy dose-volume outcome relationships. Phys Med Biol 2006;51:5719-35. [Crossref] [PubMed]
- El Naqa I, Bradley J, Blanco AI, et al. Multivariable modeling of radiotherapy outcomes, including dose-volume and clinical factors. Int J Radiat Oncol Biol Phys 2006;64:1275-86. [Crossref] [PubMed]
- Tibshirani RJ. Regression shrinkage and selection via the lasso. J R Stat Soc B 1996;58:267-88.
- Vapnik VN, Vapnik V. Statistical learning theory. Vol. 1. New York: Wiley, 1998. Available online: http://www.dsi.unive.it/~pelillo/Didattica/Artificial%20Intelligence/Old%20Stuff/Slides/SLT.pdf
- El Naqa I, Li R, Murphy MJ. editors. Machine learning in radiation oncology: theory and applications. Springer, 2015.
- Munley MT, Lo JY, Sibley GS, et al. A neural network to predict symptomatic lung injury. Phys Med Biol 1999;44:2241-9. [Crossref] [PubMed]
- Su M, Miften M, Whiddon C, et al. An artificial neural network for predicting the incidence of radiation pneumonitis. Med Phys 2005;32:318-25. [Crossref] [PubMed]
- Hastie T, Tibshirani R, Friedman JH. The elements of statistical learning: data mining, inference, and prediction: with 200 full-color illustrations. New York: Springer Google Scholar, 2001.
- Kuruvilla J, Gunavathi K. Lung cancer classification using neural networks for CT images. Comput Methods Programs Biomed 2014;113:202-9. [Crossref] [PubMed]
- Paulin F, Santhakumaran A. Back propagation neural network by comparing hidden neurons: case study on breast cancer diagnosis. Int J Comput Appl 2010;2:40-4.
- Kumar D, Wong A, Clausi DA. Lung Nodule Classification Using Deep Features in CT Images. 2015 12th Conference on Computer and Robot. Vision 2015:133-8.
- Aerts HJ, Velazquez ER, Leijenaar RT, et al. Decoding tumour phenotype by noninvasive imaging using a quantitative radiomics approach. Nat Commun 2014;5:4006. [PubMed]
- Ohri N, Duan F, Snyder BS, et al. Pretreatment 18F-FDG PET Textural Features in Locally Advanced Non-Small Cell Lung Cancer: Secondary Analysis of ACRIN 6668/RTOG 0235. J Nucl Med 2016;57:842-8. [Crossref] [PubMed]
- Naqa IE, Low DA, Bradley JD, et al. Deblurring of breathing motion artifacts in thoracic PET images by deconvolution methods. Med Phys 2006;33:3587-600. [Crossref] [PubMed]
- Aerts HJ, Grossmann P, Tan Y, et al. Defining a Radiomic Response Phenotype: A Pilot Study using targeted therapy in NSCLC. Sci Rep 2016;6:srep33860.
- Tixier F, Hatt M, Le Rest CC, et al. Reproducibility of tumor uptake heterogeneity characterization through textural feature analysis in 18F-FDG PET. J Nucl Med 2012;53:693-700. [Crossref] [PubMed]
- Cheng NM, Fang YH, Yen TC. The promise and limits of PET texture analysis. Ann Nucl Med 2013;27:867-9. [Crossref] [PubMed]
- Carles M, Torres-Espallardo I, Alberich-Bayarri A, et al. Evaluation of PET texture features with heterogeneous phantoms: complementarity and effect of motion and segmentation method. Phys Med Biol 2017;62:652. [Crossref] [PubMed]
- Oliver JA, Budzevich M, Zhang GG, et al. Variability of Image Features Computed from Conventional and Respiratory-Gated PET/CT Images of Lung Cancer. Transl Oncol 2015;8:524-34. [Crossref] [PubMed]
- El Naqa I, Kawrakow I, Fippel M, et al. A comparison of Monte Carlo dose calculation denoising techniques. Phys Med Biol 2005;50:909-22. [Crossref] [PubMed]
- Zaidi H, Abdoli M, Fuentes CL, et al. Comparative methods for PET image segmentation in pharyngolaryngeal squamous cell carcinoma. Eur J Nucl Med Mol Imaging 2012;39:881-91. [Crossref] [PubMed]
- Gunturk BK, Li X. Image restoration: fundamentals and advances. CRC Press, 2012. Available online: https://books.google.fr/books?hl=fr&lr=&id=MHPhrxEaOfcC&oi=fnd&pg=PP1&dq=Gunturk+BK,+Li+X,+eds.+Image+Restoration:+Fundamentals+and+Advances.+Boca+Raton,+FL:+CRC+Press,+Taylor+and+Francis+group%3B+2012&ots=o304Rmxg1i&sig=exo9soQB3VauQvCPteFpDrFmIXQ
占扬清
广州医科大学附属第一医院(更新时间:2021.7)
翟路路
武汉大学人民医院(更新时间:2021.7)
(本译文仅供学术交流,实际内容请以英文原文为准。)